
Meta AI разработала конкурента DeepMind AlphaFold
Исследователи Meta AI выпустили «модель белкового языка» ESM-2 с 15 млрд параметров и базу данных ESM Metagenomic Atlas, содержащую более 600 млн прогностических структур метагеномных соединений.
Белки представляют собой сложные молекулы, включающие до 20 типов аминокислот, и выполняют все виды биологических функций в организмах. Они складываются в сложные трехмерные структуры, форма которых напрямую влияет на их работу.
Определение типа соединения позволяет ученым понять принцип функционирования белков. Также данные о форме помогают им найти способы имитировать, менять или противостоять этому поведению.
Нельзя взять формулы аминокислот и сразу же определить конечную структуру, а симуляции и эксперименты отнимают много времени.
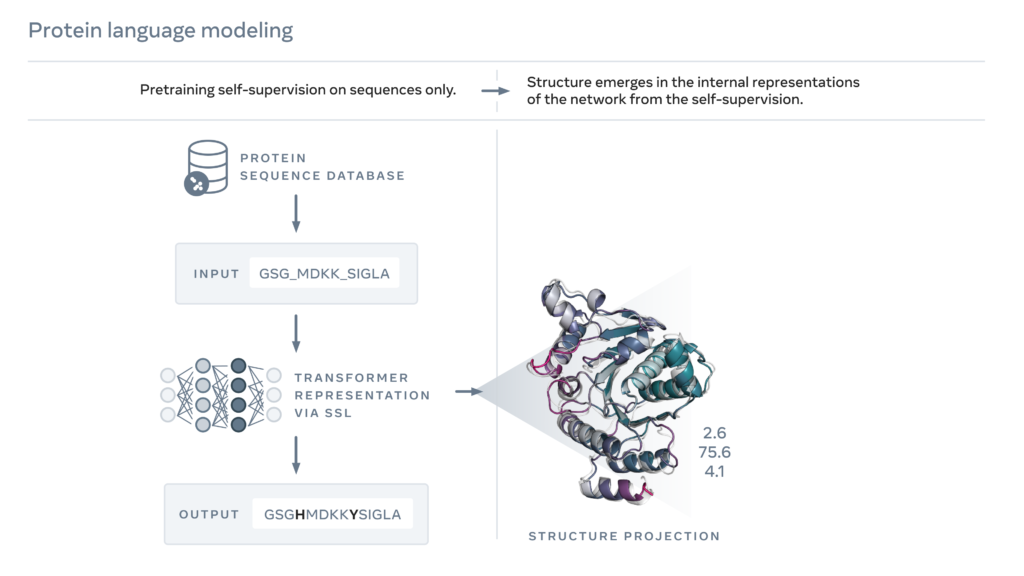
В Meta AI заявили, что нейросеть-трансформер ESM-2 представляет собой большую языковую модель, разработанную для «изучения эволюционных паттернов и создания точных прогнозов соединений непосредственно из последовательности белка».
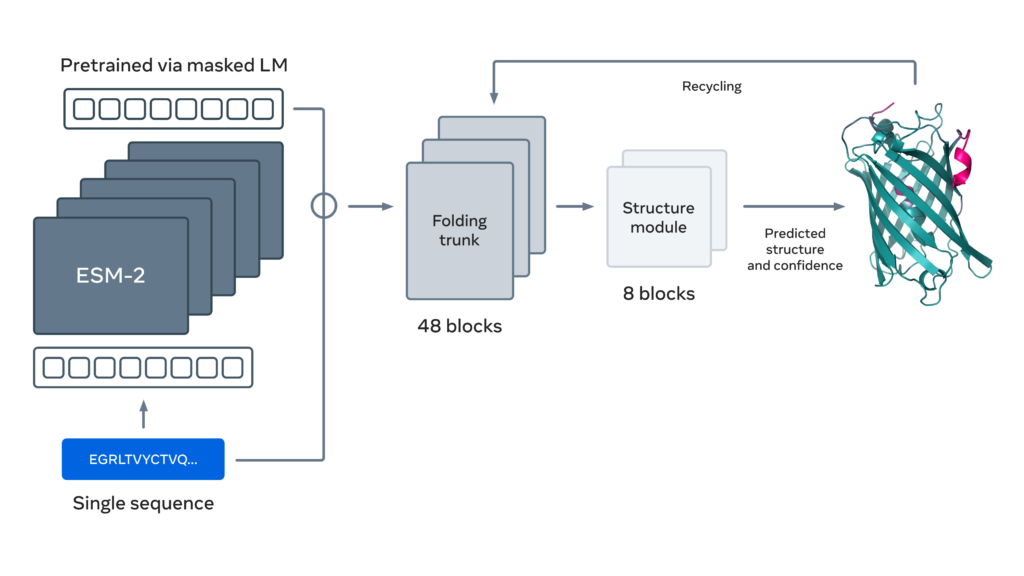
Система обрабатывает последовательности генов, используя метод самоконтролируемого обучения под названием «маскированное языковое моделирование».
По словам ученых, они натренировали алгоритм на массиве последовательностей миллионов природных белков.
«При таком подходе модель должна правильно дописывать слова в отрывке текста, например “Чтобы __ или не __, то есть __”. Мы обучили языковую модель заполнять пропуски в последовательности белков вроде “GL_KKE_AHY_G” среди миллионов различных соединений», — говорится в исследовании.
ESM-2 — самая крупная и эффективная нейросеть в своем роде. По словам ученых, алгоритм в 60 раз быстрее других современных систем вроде AlphaFold от DeepMind.
Алгоритм помог создать ESM Metagenomic Atlas, предсказав 617 млн структур из базы данных белков MGnify90 всего за две недели работы на кластере из 2000 графических процессоров. Для имитации соединения из 384 аминокислот на одной видеокарте Nvidia V100 потребуется 14,2 секунды.
«С современными вычислительными инструментами предсказание структуры сотен миллионов белков может занять годы, даже с использованием ресурсов крупного исследовательского учреждения. Чтобы делать прогнозы в масштабе метагеномики, прорыв в скорости прогнозирования имеет решающее значение», — отметили разработчики.
В Meta AI надеются, что ESM-2 и ESM Metagenomic Atlas продвинут науку и помогут специалистам, изучающим историю эволюции или борющимся с болезнями и изменением климата.
«Мы также исследуем способы применения языковых моделей для разработки новых белков и содействия решению проблем, связанных со здоровьем и окружающей средой», — добавили ученые.
Напомним, в июле алгоритм AlphaFold компании DeepMind предсказал практически все известные науке соединения, обнаруженные в растениях, бактериях и животных.
В том же месяце исследователи из MIT разработали модель глубокого обучения EquiBind, которая в 1200 раз быстрее аналогов связывает молекулы с белками при создании лекарств.
В июле 2021 года искусственный интеллект от DeepMind смоделировал 20 000 белковых структур человека.
Подписывайтесь на новости ForkLog в Telegram: ForkLog AI — все новости из мира ИИ!
Рассылки ForkLog: держите руку на пульсе биткоин-индустрии!


